RFT-Projekt: Massenspektrometrie in der Proteinforschung#
Von
Dipl.-Ing. Dr.techn. Ruth Birner-Grünberger
Univ-Prof. Dr.rer.nat. Peter Macheroux
Institut für Biochemie


Die Massenspektrometrie (MS) hat sich im letzten Jahrzehnt zu einer Schlüsseltechnologie für die naturwissenschaftliche Forschung entwickelt, deren Bedeutung für verschiedenste innovative Bereiche der Chemie, Physik und Biowissenschaften durch die Verleihung des Nobelpreises für Chemie im Jahr 2002 an John B. Fenn und Koichi Tanaka, zwei Pioniere der Massenspektrometrie, Rechnung getragen wurde. Mit Hilfe moderner Massenspektrometer lassen sich Proteine und Proteinbruchstücke auf Grund ihrer Masse und Aminosäuresequenz eindeutig identifizieren und daher stellt die Methode ein unverzichtbares Werkzeug in der Proteinforschung dar. Das vorliegende RFT-Projekt zeichnet sich durch eine starke Vernetzung mit bereits bestehenden Forschungsschwerpunkten der Technischen Universität Graz (FSP Technische Biowissenschaften) und RFT-Projekten (“Know-how cluster für Molekulares Engineering von Enzymen” und “Angewandte Biokatalyse und enzymatische Nanoanalytik”, siehe Forschungsjournal WS 2003/04) aus, die von einer verbesserten massenspektrometrischen Analytik profitieren werden.

Massenspektrometrie von Proteinen
Proteine stellen die biologisch aktiven Bausteine jeder lebenden Zelle dar und werden als lineares Polymer aus zwanzig Aminosäuren aufgebaut. Der Bauplan für alle Proteine einer Zelle ist in Form der Desoxyribonukleinsäure (DNS) gespeichert. Diese so genannte genetische Information (Erbinformation) wird in der Zelle in eine Proteinsequenz übersetzt, die sich zwangsläufig aus der zugrunde liegenden DNS-Sequenz ergibt. Daher ist es möglich bei Kenntnis der gesamten genetischen Information einer Spezies (dem Genom) die Sequenz sämtlicher Proteine und damit auch ihrer Masse zu ermitteln. Aus diesem determinierenden Zusammenhang zwischen der DNS-Sequenz des Gens und der Aminsäuresequenz des Proteins ergibt sich auch die überragende Rolle der Genomforschung (“Genomics”) für die Proteinforschung (“Proteomics”): Bei Kenntnis des Genoms erschließen sich alle Proteine eines Organismus dem Zugriff, auch solche deren biologische Funktion bislang unbekannt ist. Im Unterschied zum Genom, das vom Zeitpunkt der Entstehung eines Organismus annähernd konstant ist, ändert sich das Inventar an Proteinen (“Proteom”, die Summe der Proteine zu einem gegeben Zeitpunkt) in jedem Lebewesen in Abhängigkeit von äußeren und inneren Gegebenheiten. So reagieren Prokaryonten (Bakterien) mit der Ausbildung unterschiedlicher Proteome auf Veränderungen in ihrer Umwelt, wie z. B. die Verfügbarkeit von Nahrung, d. h. sie passen sich an ihre Umwelt an, indem bestimmte für die Situation nützliche Proteine hergestellt werden. Höhere Organismen unterliegen darüber hinaus einem inneren Entwicklungsprogramm, wie z. B. einem bestimmten determinerten Lebenszyklus, der die Bildung verschiedener Proteine zur Folge hat. Als Beispiel ist in Abb. 1 die Metamorphose eines Schmetterlings dargestellt, der für die jeweiligen Lebensphasen auf unterschiedliche Proteome zurückgreift. Dies ist ein sehr anschauliches Beispiel dafür, dass mit dem exakt gleichen Genom verschiedene Proteome dargestellt werden können, d. h. bestimmte Kombinationen von Proteinen aus den verfügbaren Genen hergestellt werden. Diese Ausprägung eines Proteoms gibt dem Organismus die Möglichkeit sich an die Umwelt anzupassen oder, wie im Beispiel des Schmetterlings dargestellt, vorprogrammierte Entwicklungsstadien zu durchlaufen.
Im Rahmen der Proteomanalytik erhält man somit Einblick in die Funktion der beteiligten Proteine und deren Zusammenwirken bei biologischen Prozessen. Daraus ergibt sich auch deren Bedeutung für die angewandte und insbesondere medizinische Forschung: Durch vergleichende Untersuchungen des Proteoms von gesunden und krankhaften Zellen lassen sich Proteine identifizieren, die möglicherweise eine Rolle bei der Entstehung der Krankheit haben oder ihre Identifizierung erweitert unser Wissen über die zugrunde liegenden molekularen Mechanismen der pathogenen Prozesse.
Experimentelles Vorgehen
Um die Möglichkeiten der Proteomforschung voll ausschöpfen zu können bedarf es zweier Voraussetzungen:
1) Verfügbarkeit einer vollständigen Genomsequenz
2) Möglichkeiten einer Auftrennung des Proteingemisches
Wie bereits zuvor erwähnt, ermöglicht die Kenntnis der gesamten DNS-Sequenz die Masse jedes Proteins einer Zelle zu bestimmen.
Darüber hinaus stehen Proteasen zur Verfügung die jedes Protein Sequenz-spezifisch nur an bestimmten Stellen zerschneiden (z. B. die Protease Trypsin spaltet ein Protein, wenn eine der beiden basischen Aminosäuren Lysin oder Arginin auftreten).
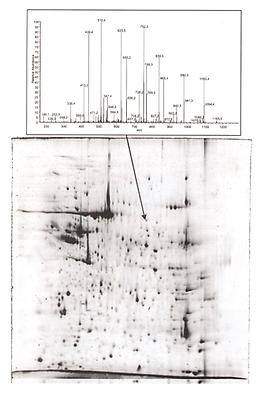
Diese Schnittstellen lassen sich ebenfalls aufgrund der vom Genom bekannten Aminosäuresequenz eines Proteins vorhersagen und daraus kann man die Masse aller Bruchstücke, die mit einer bestimmten Protease generiert werden, errechen (in silico Proteolyse, “Verdau”). Diese Peptidfragmentkarte entspricht einem so genannten peptide mass fingerprint des Proteins und dient als wesentliches Kriterium bei der Identifizierung eines Proteins.
Diese Vorgehensweise ist aber natürlich nur dann experimentell umsetzbar, wenn die Proteine aus dem vorliegenden Proteingemisch (das idealerweise die Gesamtheit aller in einer Zelle/Organismus vorhanden Proteine repräsentiert) physikalisch voneinander separiert werden können, um sie schließlich einem proteolytischen Verdau mit anschließender massenspektrometrischer Aufnahme, der für das Protein spezifischen Peptidfragmentkarte, zu unterwerfen. Diese Separation wird heute Peptidfragmentkarte mit der in silico Datenbank des betreffenden Organismus kann somit das Protein identifiziert werden. Interessant für den Proteinforscher sind natürlich diejenigen Protein spots, die in Art und Vorkommen Unterschiede erkennen lassen. So findet man z. B. in einer gesunden Zelle zum Teil andere Proteine als in einer krankhaften, worüber Aufschluss über Ursache und Entwicklung der Krankheit gewonnen werden können mit dem Ziel, diese Erkenntnisse schlussendlich für eine Therapie nutzbar zu machen.
Die Anschaffung eines matrix-assisted laser desorption/ionisation (MALDI) Massenspekrometers mit Mitteln des RFT schließt eine Lücke in der benötigten Infrastruktur für die massenspektrometrische Analytik von Biomolekülen und wird allen Arbeitsgruppen im Bereich des FSP Technische Biowissenschaften sowie dem Biokatalysezentrum Kplus für ihre zukünftige Forschung im Bereich der Proteine und Enzyme zu Gute kommen.
Weitere Informationen: www.biochemistry.tugraz.at