MOLSPEC-ID: Entwicklung von quantitativen und qualitativen molekularbiologischen Methoden zum Nachweis von Tier- und Pflanzenarten in Lebensmitteln#
Von
O.Univ.-Prof. Dr.phil. Werner Pfannhauser
Dipl.-Ing. Dr.techn. Ursula Mülleder
Institut für Lebensmittelchemie und -technologie


Das Ziel des MOLSPEC-ID-Projekts ist die Entwicklung von molekularbiologischen Detektionsmethoden zur Identifizierung spezifischer Tier- und Pflanzenarten in Lebensmitteln. Dies ist wichtig für die Aufdeckung des missbräuchlichen Ersatzes von Nahrungsmittel-Komponenten (Täuschung) und der Vermeidung von negativen Reaktionen auf unerwartete Inhaltsstoffe (Allergene).
Ausgehend von der hohen Erwartung, die von den Konsumenten in die Lebensmittelqualität gesetzt wird, ist die eindeutige Identifizierung von Tier- und Pflanzenarten in Lebensmitteln ein wichtiger Schritt in Richtung Transparenz, Information und Sicherheit. Die Lebensmittel werden immer raffinierter verarbeitet, man erkennt nicht mehr auf den ersten Blick, was alles in den Lebensmitteln enthalten ist. Das macht die Lebensmittelkennzeichnung zunehmend wichtiger. Andererseits gibt es große Qualitätsunterschiede bei den einzelnen Inhaltsstoffen und daher auch große Unterschiede bei den Produktionskosten von Lebensmitteln. Das führt zur Versuchung, Lebensmittel, die Inhaltsstoffe minderer Qualität enthalten, höherwertiger zu deklarieren als sie sind. Verfälschungen bzw. Falschdeklarationen von Fleischprodukten stellen im Vergleich zu anderen Lebensmitteln eine seltene, aber seit Jahrhunderten vorkommende Täuschung der Verbraucher dar. Der Hauptgrund für eine Falschdeklaration von Fleisch, welche durch ähnliche Eigenschaften wie Aussehen und Geschmack ermöglicht wird, liegt in der Preisdifferenz der verschiedenen Tierarten. In Zeiten von BSE ist es für viele Verbraucher wichtig zu wissen, dass als rindfleischfrei deklarierte Fleischprodukte tatsächlich kein Rindfleisch enthalten. Auch religiöse und gesellschaftliche Gründe können eine Rolle spielen, warum Konsumenten Fleisch einer bestimmten Tierart meiden. Verbraucher zahlen teilweise hohe Summen, um eine Delikatesse wie z.B. eine Gänseleberpastete zu genießen, und erwarten sich dann auch diese hohe Qualität, die nicht durch die Beimengung von Entenleber verringert sein darf. Ganz allgemein ist die Möglichkeit der Sortenidentifizierung eine Voraussetzung für die Überprüfung der Kennzeichnung von Lebensmitteln. Damit soll der Konsument vor Betrug und versteckten Lebensmittelbestandteilen, die ein großes Gesundheitsrisiko darstellen (versteckte Allergene), geschützt werden. Gerade auf dem Gebiet der potentiellen Allergene, die dafür bekannt sind, dass sie schwere anaphylaktische Reaktionen bis hin zum Tod auslösen können (Erdnuss, Soja, Sellerie), gibt es bis jetzt noch kaum validierte Nachweismethoden auf PCR (Polymerasenkettenreaktion) – Basis. Diese Methoden würden zu einer Verbesserung der Lebensmittelüberwachung und auch zur Qualitätssicherung von Rohmaterialien in der Produktion von hochqualitativen Produkten führen. Bis jetzt basieren die offiziellen Methoden zur Überprüfung von Tier- und Pflanzenarten hauptsächlich auf Proteinanalytik (z.B. isoelektrische Fokusierung, PAGE, immunologische Verfahren). Bei verarbeiteten Produkten ist das allerdings problematisch, da viele Proteine bei der Verarbeitung verändert oder zerstört werden. Deshalb haben sich in den letzten Jahren in der Lebensmittelanalytik immer mehr Methoden auf Nukleinsäure (DNA) - Basis etabliert.
Neben der hohen Empfindlichkeit und Spezifität ist die exzellente Möglichkeit der Standardisierung einer der größten Vorteile dieser Methoden. Die DNA ist in allen Zellen einer Spezies gleich. Der Informationsgehalt der DNA ist aufgrund der Degenerierung des genetischen Codes höher als der von Proteinen. DNA ist sehr stabil und übersteht die meisten Verarbeitungsschritte viel besser als Proteine. Immunologische Methoden sind immer abhängig von der hohen Qualität der Antikörper. PCR-Primer können leicht in adäquater Qualität von verschiedenen Herstellern erhalten werden. Außerdem wird zurzeit eine Vielzahl von DNA-Methoden zur Steigerung des Probendurchsatzes entwickelt (multiplex-PCR, PCR-ELISA, Chip-Technologie). Trotz allem ist auch die Proteinanalytik nicht aus dem biochemischen Alltag wegzudenken. Sie ist leicht handhabbar und relativ günstig durchführbar. Bis jetzt gibt es jedoch noch keine systematischen Vergleiche von Proteinmethoden (Limitierungen, Anwendungsbereiche) mit Methoden, die auf DNA-Basis beruhen. Ein solcher Vergleich im Rahmen des EU-Projektes MOLSPEC-ID soll neue Erfahrungswerte auf diesem Gebiet bringen und auch zu Empfehlungen in der praktischen Anwendung führen, wann z.B. welche Methode bevorzugt werden soll.
Das Hauptziel des Projektes ist die Entwicklung von Methoden, die geeignet sind für die Überwachung von potentiell allergenen Lebensmittelbestandteilen und von Ersatz von Arten und Arten- Zumischung, um eine korrekte Lebensmittelkennzeichnung zu garantieren. Dafür wird eine Reihe von Arten (tierisch und pflanzlich) in Lebensmitteln untersucht, die in dieser Beziehung besonders relevant sind. Das zu untersuchende Material wird in verschiedener Zusammensetzung und Verarbeitungsart von einem Projektpartner (der Bundesanstalt für Fleischforschung in Kulmbach in Deutschland) zur Verfügung gestellt. 14 Projektpartner aus 11 europäischen Ländern entwickeln Nachweissysteme für die verschiedenen Organismen.
Das Projekt beinhaltet zwei Aspekte:
(1) Die Entwicklung von qualitativen Methoden, die es ermöglichen,
eine breite Palette von verschiedenen Tier- und Pflanzenarten
(inklusive exotischer Tierarten und Arten von regionalem Interesse)
und versteckte potentiell allergene Stoffe nachzuweisen.
(2) Die Entwicklung von quantitativen Methoden mit besonderem Augenmerk auf die Nachweisgrenze, um die Möglichkeiten der gesetzlichen Überwachung der Lebensmittel zu verbessern.
Zusätzlich wird eine Datenbank entwickelt, in der alle Informationen, die im Rahmen des Projektes erarbeitet werden, gesammelt werden und die nach Projektabschluss dem interessierten Publikum (Lebensmittelkontrolle, Qualitätsmanagement) zur Verfügung stehen soll.
Die Arbeitsgruppe „biochemische Lebensmittelanalytik“ (Leitung Dipl.Ing. Dr. Ursula Mülleder) des Institutes für Lebensmittelchemie und –technologie der TU Graz ist aktiv in vier Arbeitsbereichen dieses EU-Projektes tätig. Im ersten Bereich geht es um die Entwicklung von qualitativen und quantitativen PCR-Methoden zur Detektion von verschiedenen Tier- und Pflanzenarten. Unsere Aufgabe ist dabei die qualitative Detektion von Truthahn, Huhn und Ente und die Entwicklung quantitativer Systeme für Ente und Truthahn. Mit den dabei erstellten Methoden wird in der Folge ein „Multiplex-PCRTUG System“ entwickelt. Dabei geht es ganz einfach um die simultane Detektion von mehreren Tierarten in einem Versuchsansatz. Der dritte Arbeitsbereich, in dem unsere Arbeitsgruppe mitwirkt, befasst sich mit dem Vergleich der neu entwickelten PCR-Methoden mit den bisher zum Nachweis von Tierarten üblichen Methoden auf Proteinbasis. Dabei werden von uns kommerziell erhältliche Testkits und Systeme anderer Projektpartner durchgetestet. Am Ende des Projektes werden schließlich einige der von den Projektteilnehmern entwickelten molekularbiologischen Methoden in Ringversuchen validiert.
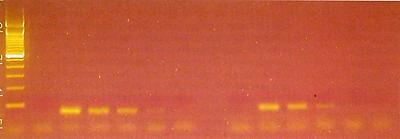
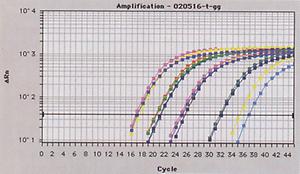
Alle von uns entwickelten Methoden beruhen auf der Polymerasekettenreaktion (PCR). Diese ermöglicht es, große Mengen an spezifischer DNA mit definierter Länge und Sequenz aus einer kleinen Menge an Ausgangs-DNA herzustellen. Dadurch können auch extrem kleine Mengen der Ziel-DNA nachgewiesen werden. Das Prinzip der PCR ist einfach. Sie umfasst 3 Schritte, wobei nach Hitzedenaturierung der DNA Oligonukleotide (Primer) an die beiden Einzelstränge anlagern und ausgehend von deren 3’-Ende die DNA-Polymerase neue DNA synthetisiert. Diese Schritte werden in Form eines Zyklus immer wieder durchlaufen, bis genug Produkt vorhanden ist (30 bis 45 Zyklen). Die Primer werden so gewählt, dass exakt nur ein kurzes DNA-Stück einer ganz bestimmten Tier- oder Pflanzenart vervielfältigt wird. Üblicherweise wird das Produkt dann mittels Agarose- Gelelektrophorese nachgewiesen. Die DNA wandert je nach Molekülgröße eine bestimmte Distanz im elektrischen Feld und kann dann mit verschiedenen Farbstoffen sichtbar gemacht werden. Durch Vergleich mit den Banden eines Größenstandards kann die Fragmentgröße bestimmt werden (Abb. 1). Mit der PCR können qualitative und semi-quantitative Nachweise durchgeführt werden. Für die quantitative Bestimmung von Truthahn und Ente werden von uns „Real-time PCR“ Methoden entwickelt. Bei der „Realtime PCR“ können die PCR-Produkte in Echtzeit gleich online durch das Messen eines Fluoreszenzsignals verfolgt werden. Es ist keine Gelelektrophorese der PCR-Produkte mehr notwendig. Je höher die Konzentration der Ziel-DNA in der Lösung ist, desto früher misst man das exponentiell ansteigende Fluoreszenzsignal (Abb.2). Mit dieser Methode können auch exakte quantitative Bestimmungen durchgeführt werden.
Am Ende des Forschungsprojektes sollen alle in diesem Rahmen entwickelten Methoden in einer Datenbank, die von einem der Projektteilnehmer erstellt wird, gesammelt vorliegen. Diese soll eine Anlaufstelle für z.B. Organe der amtlichen Lebensmittelüberwachung und andere interessierte Personen werden und zu einer weiteren Verbesserung der Kontrolle der Lebensmittelkennzeichnung und der Qualitätssicherung führen.